人工智能医学图像模型提供快速,经济高效的专家分析
加州大学洛杉矶分校的研究人员开发了一种新的人工智能模型,可以在人类临床专家所需的一小部分时间内熟练地分析疾病的3D医学图像。
这个名为SLIViT (SLice Integration by Vision Transformer)的深度学习框架分析来自不同图像模式的图像,包括视网膜扫描、超声视频、ct、核磁共振等,以识别潜在的疾病风险生物标志物。
领导这项研究的加州大学洛杉矶分校(UCLA)计算医学专家、教授埃兰·哈尔佩林博士表示,该模型在多种疾病中都非常准确,优于许多现有的针对特定疾病的基础模型。它使用了一种新的预训练和微调方法,这种方法依赖于大型的、可访问的公共数据集。因此,Halperin相信该模型可以以相对较低的成本用于识别不同的疾病生物标志物,使专家级医学成像分析客观化。
研究人员使用了NVIDIA T4 gpu和NVIDIA V100 Tensor Core gpu,以及NVIDIA CUDA来进行研究。
目前,医学图像专家经常不堪重负。在开始治疗之前,患者通常要等上几周才能接受x光、核磁共振或CT扫描评估。
SLIViT的潜在优势之一是它能够熟练地大规模分析患者数据,以及如何升级其专业知识。例如,一旦新的医学成像技术被开发出来,模型就可以根据新数据进行微调,这些数据可以被推出并用于未来的分析。
哈尔佩林博士指出,该模型也很容易部署。特别是在缺乏医学图像专家的地方,未来该模型可能会对患者的治疗结果产生重大影响。
哈尔佩林博士说,在SLIViT出现之前,以人类临床专家的水平评估大量扫描结果实际上是不可行的。有了SLIViT,大规模、准确的分析是现实的。
“该模型可以在识别疾病生物标志物方面产生巨大影响,而不需要大量手动注释的图像,”哈尔佩林博士说。“这些疾病生物标志物可以帮助我们了解患者的疾病轨迹。在未来,有可能利用这些见解来根据通过SLIVIT发现的生物标志物为患者量身定制治疗,并有望显著改善患者的生活。”
加州大学洛杉矶分校研究人员发表在《自然生物医学工程》上的一篇论文的主要作者奥伦·阿夫拉姆博士说,这项研究揭示了两个令人惊讶但又相关的结果。
两个并排的3D视频图像显示了通过光学相干断层扫描成像扫描观察到的人类视网膜的两个横截面。
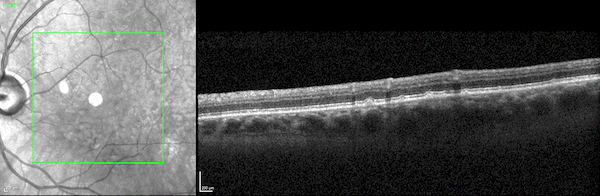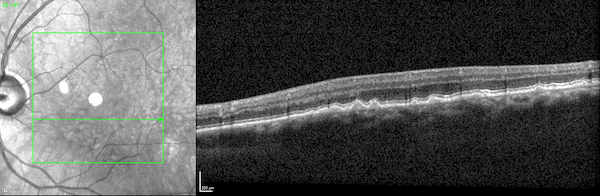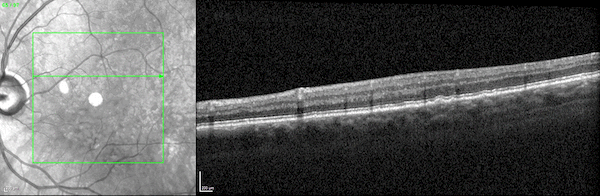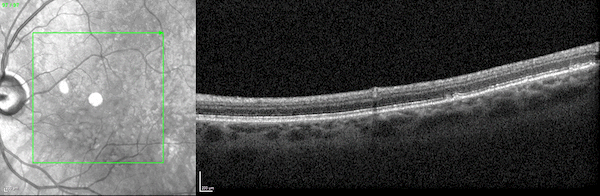
图1所示。人类视网膜的三维光学相干断层扫描
首先,虽然该模型在很大程度上是在2D扫描数据集上进行预训练的,但它能准确识别人体器官3D扫描中的疾病生物标志物。通常,设计用于分析3D图像的模型是在3D数据集上训练的。但3D医疗数据的获取成本要高得多,因此远不如2D医疗数据丰富和可访问。
加州大学洛杉矶分校的研究人员发现,通过在2D扫描上预先训练他们的模型(2D扫描更容易接近),并在相对少量的3D扫描上对其进行微调,该模型的表现优于仅在3D扫描上训练的专门模型。
第二个意想不到的结果是该模型在迁移学习方面的表现。它学会了通过微调数据集来识别不同的疾病生物标志物,这些数据集由来自不同形态和器官的图像组成。
“我们在二维视网膜扫描上训练模型,也就是眼睛的图像,然后在肝脏的核磁共振上对模型进行微调,这看起来没有联系,因为它们是两个完全不同的器官和成像技术,”Avram说。“但我们了解到,在视网膜和肝脏之间,以及在OCT和MRI之间,一些基本特征是共享的,这些特征可以用来帮助模型进行下游学习,即使图像域完全不同。”

